Allgemeines
Die „in situ“-Hybridisierung ist eine Methode, Nukleinsäuresequenzen in Geweben, Zellen, Zellkernen und Chromosomen sichtbar zu machen. Die Nukleinsäuresequenz kann direkt am biologischen Präparat „in situ“ (vor Ort) lokalisiert werden.
Bei dieser Technik bindet die Sonden-DNA an die Ziel-DNA (Patienten-DNA). Dazu müssen zuerst sowohl die Sonden- als auch die Ziel-DNA als Einzelstrang vorliegen. Das wird durch eine Hitzedenaturierung der Sonde und der DNA des zu untersuchenden Materials erzielt. Danach erfolgt eine Renaturierung, wobei sich Sonden-DNA und Patienten-DNA zusammenfügen (Bildung eines Hybrids).
Das setzt jedoch voraus, dass die Sonden-DNA komplementär zu dem DNA-Abschnitt ist, der sichtbar gemacht werden soll.
Zur Sichtbarmachung der Ziel-DNA ist die Sonden-DNA mit einem Fluoreszenz-Farbstoff gekoppelt. Zur Auswertung wird ein Fluoreszenzmikroskop genutzt.
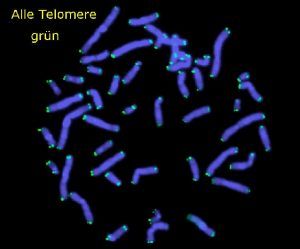
Mit der FISH-Technik ist es möglich, ein ganzes Chromosom, das Zentromer eines bestimmten Chromosoms oder aller Chromosomen, die Endbereiche der Chromosomenarme oder einzelne Abschnitte auf einem Chromosom darzustellen.